数据拟合
实验测量有时会进行审查,以便我们只知道关于特定数据点的部分信息。例如,在测量老鼠的寿命时,它们中的一部分可能在研究期间活着,因此我们只知道下限。
我们可以通过使用最大似然估计(MLE)来处理这个问题。然而,对于众所周知的分布,审查往往使分析解变得困难。
我们可以通过将MLE转换为一个凸优化问题,并使用`CVXPY <https://www.cvxpy.org/en/latest/>`__ 来解决这个挑战。
这个例子是根据 Boyd 的 CVX 101:凸优化课程 中的一道作业问题修改而来。
设置
我们将在这里使用类似的符号约定。假设我们有一个线性模型:
其中 \(y^{(i)} \in \mathbf{R}\), \(c \in \mathbf{R}^n\), \(k^{(i)} \in \mathbf{R}^n\), 而 \(\epsilon^{(i)}\) 是误差,其服从正态分布 \(N(0, \sigma^2)\),其中 $ i = 1,,K$。
那么MLE估计 \(c\) 就是使得误差平方和 \(\epsilon^{(i)}\) 最小的向量,即:.. math:
\begin{array}{ll}
\underset{c}{\mbox{最小化}} & \sum_{i=1}^K (y^{(i)} - c^T x^{(i)})^2
\end{array}
对于右截尾数据的情况下,只有 \(M\) 个观测是完全观测到的,并且对于其余的观测只知道 \(y^{(i)} \geq D\), 其中 \(i=\mbox{M+1},\ldots,K\) 且 \(D\) 是一个常数。
现在让我们看看在实践中如何工作。
数据生成
import numpy as np
n = 30 # 变量数量
M = 50 # 截尾观测数量
K = 200 # 总观测数量
np.random.seed(n*M*K)
X = np.random.randn(K*n).reshape(K, n)
c_true = np.random.rand(n)
# 生成 y 变量
y = X.dot(c_true) + .3*np.sqrt(n)*np.random.randn(K)
# 基于 y 进行排序
order = np.argsort(y)
y_ordered = y[order]
X_ordered = X[order,:]
# 寻找边界
D = (y_ordered[M-1] + y_ordered[M])/2.
# 应用截尾
y_censored = np.concatenate((y_ordered[:M], np.ones(K-M)*D))
import matplotlib.pyplot as plt
# 在 ipython 中内联显示图表。
%matplotlib inline
def plot_fit(fit, fit_label):
plt.figure(figsize=(10,6))
plt.grid()
plt.plot(y_censored, 'bo', label = '截尾数据')
plt.plot(y_ordered, 'co', label = '未截尾数据')
plt.plot(fit, 'ro', label=fit_label)
plt.ylabel('y')
plt.legend(loc=0)
plt.xlabel('观测值');
普通最小二乘法(OLS)
让我们看看普通最小二乘法(OLS)的结果如何。我们将使用``np.linalg.lstsq``函数来求解系数。
c_ols = np.linalg.lstsq(X_ordered, y_censored, rcond=None)[0]
fit_ols = X_ordered.dot(c_ols)
plot_fit(fit_ols, 'OLS fit')
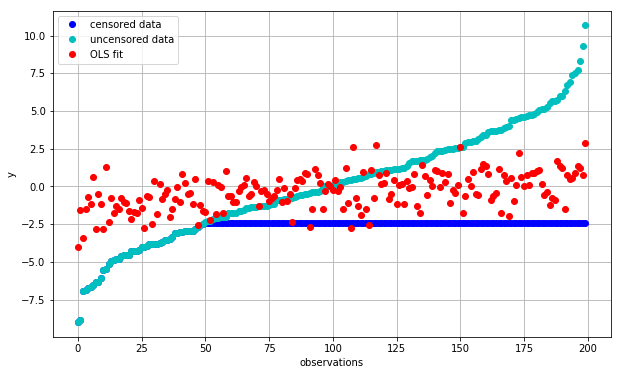
我们可以看到,我们在低于真实值下估计 \(y\) ,反之亦然(红色与青色)。 这是由于使用了有审查(蓝色)的观察结果,这些观察结果对趋势线产生了很大的影响, 将其拉向红色和蓝色点之间的误差。
使用无审查数据的OLS
为了保持分析的可处理性,最简单的方法是简单地忽略所有审查观测结果。
考虑到我们的 \(M\) 远小于 \(K\) ,我们为了做到这一点而扔掉 了大多数数据集,让我们看看这个新的回归如何工作。

Now, by incorporating these additional constraints, we can see that the predictions are no longer below \(D\) for the censored portion. The overall fit has improved and it properly takes into account the boundaries of the censored data.
import cvxpy as cp X_uncensored = X_ordered[:M, :] c = cp.Variable(shape=n) objective = cp.Minimize(cp.sum_squares(X_uncensored*c - y_ordered[:M])) constraints = [ X_ordered[M:,:]*c >= D] prob = cp.Problem(objective, constraints) result = prob.solve()
c_cvx = np.array(c.value).flatten() fit_cvx = X_ordered.dot(c_cvx) plot_fit(fit_cvx, ‘CVX fit’)
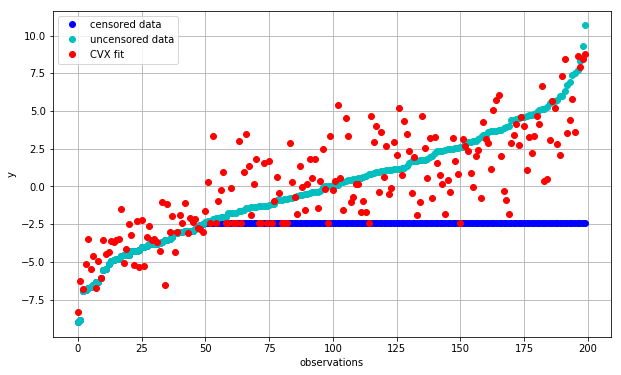
从质量上看,这个拟合结果已经比之前好了,因为它不再对未被审查的部分预测不一致的值。但是它是否能够找到与原始数据接近的系数 \(c\) 呢?
我们将使用简单的欧氏距离 \(\|c_\mbox{true} - c\|_2\) 来进行比较:
print("norm(c_true - c_cvx): {:.2f}".format(np.linalg.norm((c_true - c_cvx))))
print("norm(c_true - c_ols_uncensored): {:.2f}".format(np.linalg.norm((c_true - c_ols_uncensored))))
norm(c_true - c_cvx): 1.49
norm(c_true - c_ols_uncensored): 2.23
结论 ———-将带有参数分布的被审查数据进行拟合往往是具有挑战性的,因为最大似然估计(MLE)的解往往无法通过解析计算得到。然而,许多MLE问题可以转化为凸优化问题,如上所示。随着易于使用且健壮的数值计算软件包的出现,我们现在可以轻松地解决这些问题,同时在数据的各个部分上强制执行一致性条件,以考虑到我们的全部信息集合。